|

Englische Version
Site map
FAIR 3889
Viren
Phytoplasmen
Pathogensammlung
Pathogennachweis
ACLSV
ApMV
ASGV
ASPV
PPV
PDV
PNRSV
ArMV
ToRSV
RpRSV
SLRSV
GFLV
GLRaV-1
GLRaV-3
LChV
CMLV
CRMV
CNRMV
CGRMV
ChTLV
CVA
AP
ESFY
PD
Pathogeneliminierung
Kontakt
Nützliche Links
|
Pathogennachweis
Zum Nachweis von Pathogenen stehen verschiedene Methoden zur Verfügung.
Die Methodenwahl hängt vom nachzuweisenden Pathogen, der erwünschten
Schnelligkeit, vorhandenen Ausrüstung und dem finanziellen Rahmen ab.
Alle diese Aspekte sollten Berücksichtigung finden, wenn ein Testsystem
für kommerziell gehandeltes Vermehrungsmaterial ausgewählt wird.
International zugelassene Nachweissysteme schließen serologische und
molekulare Labormethoden sowie Indexierung im Glashaus und Freiland ein. Sie
werden von der Arbeitsgruppe für Obstvirosen der ISHS geprüft und
im Rahmen regelmäßiger Treffen (Wien 1991 (Acta Hort. 309: 407-418,
1992), Bethesda 1997 (Acta Hort. 472: 761-783, 1998), Canterbury 2000)
auf neuestem Stand gehalten.
Eine Übersicht dieser Methoden für Kernobst und
Steinobst findet sich in Tabellenform am Ende dieser
Seite.
Viren
Viren können durch
Indexing im Freiland und Glashaus, durch serologische
Methoden, bildgebende Verfahren wie beispielsweise Elektronenmikroskopie,
molekulare Hybridisierung, Nucleic Acid Sequence Based Amplification (NASBA)
und PCR nachgewiesen werden.
Die ISHS ´Working Group on Virus Diseases of Fruit Trees´
empfiehlt serologisches Indexing (ELISA) seit vielen Jahren. Seit der
Konferenz von Bethesda im Jahre 1997 werden vor allem Anstrengungen unternommen,
Testsysteme auf der Basis der PCR zu entwickeln, die sich durch hohe
Sensitivität und Geschwindigkeit auszeichnen.
Im Rahmen des Projekts FAIR 3889 wurden sowohl für RNA-Präparation
wie auch für breitbandigen und spezifischen Nachweis von Obstvirosen
verbesserte Verfahren entwickelt.
Zum Seitenanfang
Phytoplasmen
Da Phytoplasmen charakteristische morphologische Anomalien verursachen, können
sie durch genaue Inspektion von Obstanlagen und Baumschulen erkannt werden.
Zudem ist ein zuverlässiger Nachweis von ESFY durch Indexing auf
GF 305-Unterlagen im Glashaus möglich, wobei Inkubationszeiten von
4 Monaten abzuwarten sind. In den Siebröhren von Blattstielen, Borke
und Wurzeln können Phytoplasmen durch
DAPI-Färbung
sichtbar gemacht werden.
Der Nachweis mittels PCR als rasches und zuverlässiges Diagnoseverfahren
wurde im Rahmen des Projekts FAIR 3889 optimiert. Die Verlässlichkeit dieses
Nachweisverfahrens hängt entscheidend von der Qualität der eingesetzten
DNA ab. Aus diesem Grunde wurden verschiedene Protokolle zur DNA-Reinigung
untersucht. Durch die Verfahren der Plate Capture PCR und der IC-PCR
kann zudem der initiale Reinigungsschritt durch die Verwendung von Antisera
entscheident vereinfacht werden.
Zum Seitenanfang
Indexierung
Indexierung basiert auf der Fähigkeit bestimmter Indikatorpflanzen,
nach Infektion mit einem Virus typische Krankheitsmerkmale zu entwickeln.
Die zu testende Sorte wird auf die Indiktorpflanze veredelt oder es wird eine
mechanische Übertragung von Pflanzensaft durchgeführt. Nach einer
festgelegten Inkubationszeit werden die Indikatorpflanzen auf das
Vorhandensein von sichtbaren Symptomen untersucht.
Die folgende Tabelle gibt eine Übersicht emfohlener Indexierungsverfahren,
der verwendeten Indikatorpflanzen sowie der Viren, die nachgewiesen werden
können.
Weitere Informationen zum Thema Indexing finden sie auf der Homepage
der Washington
State University.
| Typ | Indikator | Zeitaufwand |
Nachweisbare Viren |
|---|
| Glashausindexierung mit krautigen Indikatoren |
Chenopodium quinoa | 20 Tage |
ACLSV, ASGV, Nepoviren |
| Cucumis sativus | 20 Tage |
ApMV, PDV, PNRSV |
| Glashausindexierung mit holzigen Indikatoren |
Malus platycarpa | 8 Wochen |
ACLSV |
| Malus pumila ´Virginia Crab´ | 24 Wochen |
ASGV, ASPV |
| Malus pumila R 12740 7A | 4 Wochen |
ACLSV |
| Malus pumila spy 227 | 12 Wochen |
ASPV |
| Cydonia oblonga C 7/1 | 5 Wochen |
ACLSV |
| Prunus persica GF 305 | 8 Wochen |
ACLSV, PPV, PDV, PNRSV, SLRSV, ESFY |
| Prunus tomentosa | 12 Wochen |
ACLSV, PPV, PDV, PNRSV |
| Prunus serrulata ´Shirofugen´ | 8 Wochen |
PDV, PNRSV |
| Freilandindexierung |
Malus platycarpa | 2 Jahre |
ACLSV |
| Pyronia veitchii | 2 Jahre |
ASPV |
| Malus pumila ´Virginia crab´ | 3 Jahre |
ASGV, ASPV |
| Malus pumila R 12740 7A | 2 Jahre |
ACLSV |
| Malus pumila spy 227 | 2 Jahre |
ASPV |
| Malus pumila ´Lord Lambourne´ | 3 Jahre |
ApMV, rubbery wood, flat limb, chat fruit |
| Malus pumila ´Gravensteiner´ | 3 Jahre |
flat limb |
| Malus pumila ´Golden Delicious´ | 2 Jahre |
ApMV, AP |
| Prunus serrulata ´Shirofugen´ | 6 Wochen -
2 Jahre |
PDV, PNRSV |
| Prunus serrulata ´Kwanzan´ | 2 Jahre |
CGRMV |
| Prunus avium ´Bing´ | 3 Jahre |
CRLV, CTLV, SLRSV, ArMV, CNRSM, CRMV |
| Prunus avium ´Sam´ | 3 Jahre |
LChV |
| Prunus avium Canindex I | 3 Jahre |
CNRMV |
| Prunus persica GF 305 | 4 Jahre |
ACLSV, PPV, PDV, PNRSV, ESFY |
Zum Seitenanfang
Serologische Methoden
Der ELISA-Test (Enzyme Linked Immunosorbant Assay) wurde bereits vor über
30 Jahren zum Nachweis von
Pflanzenviren entwickelt. Er wird routinemässig zur Massenuntersuchung
von Pflanzen eingesetzt, ist rasch, kostengünstig und einfach
durchzuführen. Allerdings kann er nur für Pathogene eingesetzt
werden, von denen Antiseren im Handel erhältlich sind.
Die Einsetzbarkeit wird weiters durch die ungleiche Verteilung mancher
Pathogene in der Pflanze sowie durch klimatische Einflüsse, die den
Virustiter unter die Nachweisgrenze drücken können, eingeschränkt.
Nach der Anwendung von Verfahren zur Viruseliminierung muss berücksichtigt
werden, dass es Monate dauern kann, bis eventuell verbliebene Viren wieder
einen mittels ELISA nachweisbaren Titer aufgebaut haben.
Die Methode des Immuno-Tissue-Printing ermöglicht die Erfassung der
Virusverteilung im Pflanzengewebe und kann damit zur Verbesserung von
in vitro Eliminierungsverfahren beitragen.
Zum Seitenanfang
Polymerase-Kettenreaktion (PCR)
Mit der Polymerase-Kettenreaktion können spezifische DNA-Abschnitte
so stark vermehrt werden, dass ihr Nachweis anschliessend leicht
möglich ist. Diese DNA wird üblicherweise durch Gel-Elektrophorese
sichtbar gemacht.
Im Pathogennachweis hat sich die PCR um einen Faktor 100 bis 1000 sensitiver
als der ELISA-Test erwiesen. Daher ist sie besonders für den Nachweis
geringer Virenkonzentrationen, wie sie vor allem bei frischen Infektionen
auftreten, von Nutzen.
Zahlreiche Seiten im World Wide Web beschäftigen sich mit der PCR.
Die folgenden Links geben einen kurzen technischen Überblick über diese
Methode:
"What the heck is PCR" von C. Brown
"Principle of PCR" von Andy Vierstraete
Kurze Beschreibung der PCR von der University of Califormia
Detaillierte PCR-Protokolle vom University College in London
Die DNA von Phytoplasmen kann direkt in der PCR eingesetzt werden.
Die meisten Pflanzenviren enthalten hingegen RNA als Erbsubstanz. Für
deren Nachweis ist es erforderlich, diese RNA erst zu reinigen und dann
mit Hilfe eines Enzyms (RT, reverse Transkriptase) in DNA zu übersetzen,
die dann mittels PCR vermehrt werden kann.
Zum Pathogennachweis werden verschiedene Primer-Paare verwendet. Dabei handelt
es sich um kurze Fragmente aus der Erbsubstanz des Pathogens, die die
vermehrten Abschnitte flankieren. Im Rahmen des Projekts FAIR 3889 wurden
Primerpaare für verschiedene bedeutende Obstvirosen entwickelt.
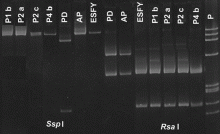
Die PCR-Produkte können durch enzymatischen Verdau weiter untersucht werden.
Dadurch erhält man ´Fingerabdrücke´, die einerseits
den Virusnachweis zusätzlich absichern, andererseits verwendet werden
können, um Untergruppen bestimmter Viren zu identifizieren.
Zum Seitenanfang
Serologische und molekulare Labormethoden zum
Nachweis von Viren und Phytoplasmen in Kernobst
Pathogen
(für Details anklicken) |
Serologische Tests | Molekulare Tests | Literatur |
|---|
| ACLSV | ELISA |
RT-PCR
IC-RT-PCR
RT-PCR ELOSA |
6, 27, 30, 31, 41 |
| ASGV | ELISA |
RT-PCR
IC-RT-PCR
RT-PCR ELOSA |
9, 27, 29, 30, 31, 37, 39 |
| ASPV | |
RT-PCR
NASBA |
25, 29, 30, 31, 38, 40, 58 |
| ApMV | ELISA |
RT-PCR
IC-RT-PCR
RT-PCR ELISA |
5, 56 |
| ToRSV | ELISA |
RT-PCR
RT-PCR ELISA
Hybridization |
18, 19, 43, 56 |
| AP | ELISA |
PCR
IC-PCR |
8, 21, 35, 36, 59 |
| PD | |
PCR |
8, 10, 12, 35, 36, 59 |
Zum Seitenanfang
Serologische und molekulare Labormethoden zum
Nachweis von Viren und Phytoplasmen in Steinobst
Pathogen
(für Details anklicken) |
Serologische Tests | Molekulare Tests | Referenz |
|---|
| ACLSV | ELISA |
RT-PCR
IC-RT-PCR |
6, 27, 30, 31, 41 |
| ApMV | ELISA |
RT-PCR
IC-RT-PCR
RT-PCR ELISA |
5, 56 |
| PPV | ELISA |
RT-PCR
IC-RT-PCR |
|
| PDV | ELISA |
RT-PCR
IC-RT-PCR |
42 |
| PNRSV | ELISA |
RT-PCR
RT-PCR ELISA |
5, 20, 60 |
| ToRSV | ELISA |
RT-PCR
RT-PCR ELISA
Hybridization |
18, 19, 43, 56 |
| ArMV | ELISA |
RT-PCR
Hybridization
NASBA |
19, 37 |
| RpRSV | ELISA |
NASBA |
|
| SLRSV | ELISA |
NASBA |
|
| LChV | |
RT-PCR |
26, 28, 46, 49, 50, 53, 62 |
| CMLV | ELISA |
|
|
| CRLV | ELISA |
|
|
| CVA | |
RT-PCR
IC-RT-PCR |
22 |
| TRSV | ELISA |
|
|
| TBRV | ELISA |
|
|
| ESFY | |
PCR |
1, 8, 11, 13, 21, 23, 32, 35, 59 |
Zum Seitenanfang
Literatur
- Ahrens U., Lorenz K. H., and Seemüller E. 1993. Genetic diversity among
mycoplasmalike organisms associated with stone fruit diseases. Molecular
Plant-Microbe Interactions, 6: 686-691.
- Bertaccini A., Carraro L., Davies. D.L., Laimer da Câmara Machado M.,
Martini M., Paltrinieri S., and Seemüller E., 2000. Micropropagation of a collection
of phytoplasma strains in periwinkle and other host plants. 13th International
Congress of IOM, ACROS Fukuoka, Japan, July 14-19 2000: 101.
- Bertaccini A., L. Carraro, D. Davies; M. Laimer da Câmara Machado, M.
Martini, S. Paltrinieri, and Seemüller E., 2000. Micropropagation of a collection
of phytoplasma strains in periwinkle and other host plants. EFPP 2000,
Taormina-Giardini Naxos, 19-22 September 2000, 122P: 38.
- Candresse, T., 2001. Advances in the methods of pathogen detection.
Acta Hort. accepted.
- Candresse T., Kofalvi S. A., Lanneau M., and Dunez J., 1998. A PCR-ELISA
procedure for the simultaneous detection and identification of prunus necrotic
ringspot and apple mosaic ilarviruses. Acta Hort. 472.
- Candresse T., Lanneau M., Revers F., Grasseau N., Macquaire G., German S.,
Malinowsky T., and Dunez J., 1995. An immunocapture PCR assay adapted to the
detection and the analysis of the molecular variability of the apple chlorotic
leafspot virus. Acta Hort. 386: 136-147.
- Candresse, T., Lanneau, M., Revers, F., Kofalvi, S. and Macquaire, G., 2000.
PCR-based techniques for the detection of plant viruses and viroids.
Acta Hort. 530: 61-67.
- Carraro L., Nemchinov L., and Hadidi A., 1998. PCR detection of pome and
stone fruit phytoplasmas from active or dormant tissue. Acta Hort.
472: 731-735.
- Crossley S. J., Jacobi V., and Adams A. N., 1998. IC-PCR amplification of
apple stem grooving virus isolates and comparison of polymerase and coat protein
gene sequences. Acta Hort. 472.
- Davies D.L., Clark M.F. and Adams A.N. 1998. The epidemiology of Pear decline
in the UK. Acta Hort. 472: 669-672.
- Davies DL, and Adams AN., 2000. European stone fruit yellows phytoplasmas
associated with a decline disease of apricots in southern England. Plant Pathology
(in press).
- Davies DL., and Clark MF., 1994. Maintenance of MLOs occurring in Pyrus species
by micropropagation and their elimination by tetracycline therapy. Plant
Pathology 43: 819 - 823.
- Davies D. L., and Clark M. F., 1992. Production and characterisation of
polyclonal and monoclonal antibodies against peach yellow leafroll MLO-associated
antigens. Acta Hort. 309: 275-283.
- Foissac X., Svanella-Dumas L., Gentit P., Dulucq M.J., and Candresse T., 2001.
Polyvalent detection of fruit tree tricho, capillo and foveaviruses by nested
RT-PCR using degenerated and inosine containing primers (PDO-RT-PCR)
Acta Hort. accepted.
- Gentit P., Delbos R.P., Candresse T., and Dunez, J., 2001. Characterisation of
a new Nepovirus infecting apricot in the Southeastern France: Apricot latent
ringspot virus. Eur. J. Plant Pathol. accepted.
- Gentit P., Foissac X., Svanella-Dumas L., Peypelut M., and Candresse, T., 2001.
Biological properties and molecular characterization of two different Foveaviruses
inducing similar disorders in cherry trees. Acta Hort. accepted.
- Gentit P., Foissac X., Svanella-Dumas L., and Candresse T., 2001. Variants of
Apricot latent foveavirus (ALV) isolated from south european orchards associated
with peach asteroid spot and peach sooty ringspot. Acta Hort. accepted.
- Griesbach J. A., 1995. Detection of tomato ringspot virus by polymerase
chain reaction. Plant Dis. 79: 1054-1056.
- Hadidi A., and Hammond R. W., 1989. Construction of molecular clones for
identification and detection of tomato ringspot and arabis mosaic viruses. Acta
Hort. 235: 223-230.
- Hammond R., Howell W. E., Mink G. I., and Crosslin J. M., 1998.
Strain-specific polymerase chain reaction assays for discrimination of prunus
necrotic ringspot virus isolates. Acta Hort. 472.
- Heinrich M., Simona Botti, Licia Caprara, Arthofer W., Strommer S., Hanzer V.,
Paltrinieri S., Martini M., Katinger H. Bertaccini A., and Laimer da Câmara
Machado M., 2001. Development and evaluation of improved detection methods for
phytoplasmas in fruit tree. In preparation.
- James D., and Jelkmann W., 1998. Detection of cherry virus A in Canada
and Germany. Acta Hort. 472.
- Jarausch W., Lansac M., Saillard C., Broquaire J.M. and Dosba F., 1998. PCR
assays for specific detection of European stone fruit yellows phytoplasmas and its
use for epidemiological studies in France. European Journal of Plant Pathology
104: 17-27.
- Jelkmann W., 1998. Identification and detection of recalcitrant temperate
fruit crop viruses using dsRNAs and diffusion antisera. In: Plant Virus
Disease Control, A. Hadidi, R. K. Khetarpal, and H. Koganezawa, eds. APS
Press, St. Paul, MN, pp. 392-398.
- Jelkmann W., and Keim-Konrad R., 1997. An immuno-capture polymerase chain
reaction and plate-trapped ELISA for the detection of apple stem pitting virus.
J. Phytopathol. 145: 499-504.
- Jelkmann W., Keim-Konrad R., Vitushkina M., and Fechtner B., 1998.
Complete nucletotide sequences of little cherry closterovirus and virus
detection by polymerase chain reaction. Acta Hort. 472.
- Kinard G. R., Scott S. W., and Barnett O. W., 1996. Detection of apple
chlorotic leaf spot and apple stem grooving viruses using RT-PCR.
Plant Dis. 80: 616-621.
- Kountrias A., Eppler A., Rott M. E., Jelkmann W., and Adam, G. (2000).
Preliminary field results on two causal agents of little cherry disease in the
fruit growing area 'Altes Land' of Northern Germany. Acta Hort. (in press).
- Kummert J., Marinho V.L.A., Rufflard G., Colinet D., and Lepoivre P., 1998.
Sensitive detection of apple stem grooving and apple stem pitting viruses from
infected apple trees by RT-PCR. Acta Hort. 472: 97-104.
- Kummert J., Vendrame M., Steyer S., and Lepoivre P., 2000. Development of
routine RT-PCR tests for certification of fruit tree multiplication material.
EPPO Conference on Diagnostic Techniques for Plant Pests (Wageningen,
NL - 2000-02-1/4) Bulletin OEPP/EPPO Bulletin, 30.
- Kummert J., Vendrame M., Lepoivre P., and Steyer S., 2001. Development of
routine RT-PCR ELOSA tests for fruit tree certification. 18th International
Symposium on virus-like didease of temperate fruit crops - ISHS (Canterbury,
UK - 2000-07-9/15) Acta Hort. in press
- Laimer da Câmara Machado M., Paltrinieri S., Hanzer V., Arthofer W.,
Strommer S., Martini M., Pondrelli M. and Bertaccini A., 2001. Presence of
European stone fruit (ESFY or 16SrX-B) phytoplasmas in apricots in Austria.
Plant Pathology 50 (1) 130 - 135.
- Laimer da Câmara Machado M., Heinrich M., Hanzer V., Arthofer W.,
Strommer S., Paltrinieri S., Martini M., Bertaccini A., Kummert J. and Davies
D.L. 2001. Improved detection of viruses and phytoplasmas in fruit tree tissue
cultures. 18th ISHS Conference on Virus and Virus-like Diseases of Temperate
Fruit Crops. Canterbury, 9. - 15. July 2000. Acta Hort. accepted. in press
- Laimer da Câmara Machado M., Kummert J., Candresse T., Jelkmann W.,
Cassells A., Bertaccini A., van den Heuvel J.F.J.M., Davies. D.L. 1999. Health
certification of rosaceous species based on disease-indexing of in vitro plants:
validation of diagnostics and diagnostic strategies. ISHS Conference on Methods
and Markers for Quality Assurance in Micropropagation. Cork. 24-27 August 1999.
Abstracts: 104-106.
- Lee I.M., Bertaccini A., Vibio M. and Gunderson D.E., 1995. Detection of
multiple phytoplasmas in perennial fruit trees with decline symptoms in Italy.
Phytopathol. 85: 728-735.
- Lorenz K.-H., Schneider B., Ahrens U., and Seemüller E., 1995.
Detection of apple proliferation and pear decline phytoplasmas by PCR
amplification of ribosomal and nonribosomal DNA. Phytopathol. 85: 771-776.
- MacKenzie D. J., McLean M. A., Mukerji S., and Green M., 1997. Improved
RNA extraction from woody plants for the detection of viral pathogens by
reverse transcription-polymerase chain reaction. Plant Dis. 81: 222-226.
- Malinowski T., Komorowska B., Gokis T., and Zawadzka B., 1988.
Detection of apple stem pitting virus and pear vein yellows virus using
reverse transcription - polymerase chain reaction. Acta Hort. 472.
- Marinho V.L.A., Kummert J., Rufflard G., Colinet D., and Lepoivre P., 1998.
Detection of apple stem grooving virus in dormant apple trees by using crude
extracts as templates for one-step-RT-PCR. Plant Diseases 82: 785-790.
- Nemchinov L., Hadidi A., and Faggioli F., 1998. PCR-detection of apple
stem pitting virus from pome fruit hosts and sequence variability among
viral isolates. Acta Hort. 472.
- Nemchinov L., Hadidi A., Candresse T., Foster J. A., and Verderevskaya T.,
1995. Sensitive detection of apple chlorotic leaf spot virus from infected
apple or peach tissue using RT-PCR, IC-RT-PCR, or multiplex IC-RT-PCR.
Acta Hort. 386: 51-62.
- Parakh DR., Shamloul AM., Hadidi A., Waterworth HE., Scott SW., Howell HE.
and Mink G., 1995. Detection of Prune Dwarf Virus from infected stone fruits using
reverse transcription polymerase chain reaction. Acta Horti. 386: 421 - 430.
- Powell C. A., Hadidi A., and Halbrendt J. M., 1991. Detection and
distribution of tomato ringspot virus in infected nectarine trees using
ELISA and transcribed RNA probes. HortScience 26: 1290-1292.
- Rott M., and Jelkmann W., 1999. Detection of filamentous viruses from sweet
cherry. Phytomedizin 1, 48.
- Rott M., and Jelkmann W., 1999. Detection and characterization of several
filamentous viruses from sweet cherry. XIth International Congress of Virology
9-13 Aug. 1999, 368.
- Rott M., and Jelkmann W., 1999. Charakterisierung, PCR-Diagnose und
Untersuchungen zur Verbreitung eines zweiten Closterovirus als Verursacher der
litte cherry Erkrankung an Süßkirschen. Phytomedizin 3, 64.
- Rott M. E., and Jelkmann W., 2000. Complete nucleotide sequence of cherry
necrotic rusty mottle virus. Archives of Virology (in press).
- Rott M. E., and Jelkmann W., 2000. Complete nucleotide sequence of cherry
necrotic mottle virus. Phytopathol. 90, 67.
- Rott M. E., and Jelkmann W., 2000. Detection and partial characterization of a
second viral agent associated with little cherry disease. Phythopthology 90, 67.
- Rott M. E., and Jelkmann W., 2000. Detection and partial characterization of
a second viral agent associated with little cherry disease. Acta Hort. (in press).
- Rott M. E., and Jelkmann W., 2000. Complete nucleotide sequence of cherry
necrotic rusty mottle virus. Acta Hort. (in press).
- Rott M. E., and Jelkmann W., 2000. Development of PCR primer pairs for the
characterization and detection of several related filamentous viruses of cherry.
Acta Hort. (in press).
- Rott M. E., and Jelkmann W., 2000. Molekulare Charakterisierung eines zweiten
Closterovirus assoziiert mit der Kleinfrüchtigkeit der Sübkirsche (little cherry).
Mitteilungen aus der Biologischen Bundesanstalt für Land- und Forstwirtschaft
Berlin-Dahlem (im Druck).
- Rott M. E., and Jelkmann W., 2000. Neue Erkenntnisse und Entwicklung von
Nachweisverfahren für wirtschaftlich bedeutsame und wenig beschriebene Kirschvirosen.
Mitteilungen aus der Biologischen Bundesanstalt für Land- und Forstwirtschaft
Berlin-Dahlem (im Druck).
- Rott M. E., and Jelkmann W., 2000. Charakterisierung und Nachweis filamentöser
Viren an Kirschen und Verwandtschaften zu cherry green ring mottle virus.
Phytomedizin 2, 25.
- Rowhani A., Biardi L., and Golino D. A., 1998. Detection of viruses of woody
host plants using colorimteric PCR. Acta Hort. 472.
- Rowhani A., Maningas M. A., Lile L. S., Daubert S. D., and Golino D. A.,
1995. Development of a detection system for viruses of woody plants based
on PCR analysis of immobilized virions. Phytopathol. 85: 347-352.
- Schwarz K., and Jelkmann W., 1998. Detection and characterization of
European apple stem pitting virus isolates of apple and pear by PCR and
partial sequence analysis. Acta Hort. 472.
- Smart C.D., Schneider B., Blomquist C.L., Guerra L.J., Harrison N.A.,
Ahrens U., Lorenz K.H., Seemuller E. and Kirkpatrick B.C., 1996. Phytoplasma
specific PCR primers based on sequences of the 16S - 23S rRNA spacer region.
Applied and Environmental Microbiology 62: 2988-2993.
- Spiegel S., Scott S. W., Bowman-Vance V., Tam Y., Galiakparov N. N., and
Rosner A., 1996. Improved detection of prunus necrotic ringspot virus by the
polymerase chain reaction. Eur. J. Plant Pathol. 102: 681-685.
- Veronesi F., Bertaccini A., Parente A., Mastronicola M., and Pastore M., 2000.
PCR indexing of phytoplasma-infected micropropagated periwinkle treated with
PAP-II, a ribosome inactivating protein from Phytolacca americana leaves.
Acta Hort. 530: 113-119.
- Vitushkina M., Fechtner B., AgranovskyA. A., and Jelkmann W., 1997.
Development of an RT-PCR for the detection of little cherry virus and
characterization of some isolates occuring in Europe. Eur. J. Plant Pathol.
103: 803-808.
Back to pagetop
|